FIDA技术的独到之处在于,在溶液中仅需少量样品,能够在纳米尺度上检测分子及其复合物的大小尺寸(size)和变化,检测所得为绝对值,准确可靠(可参考下文中的实验数据;往期内容也可见其数据与小角度X光散射法SAXS的结果相一致)。而分子和分子复合物尺寸的大小和变化(size changes)是反映其结构的一个重要参数,而对一些生物大分子如蛋白质而言,其生物学功能就需要建立在相应的三维结构之上。集成入蛋白结构数据库(PDB)的Fida 1系统,如虎添翼,可将检测所得的流体力学半径(Rh)与数据库中X射线、冷冻电镜(Cryo-EM)的结构数据,以及AlphaFold预测所得的结构数据予以精确关联,建立了End-to-end定量的工作流程。
简介
在生物物理学中,分子尺寸大小的变化(size changes)是蛋白和其复合物结构的一个重要的定量参数。若要将蛋白结构与其尺寸大小(如Rh,流体力学半径,也称为水力学半径)关联,需要基于实验或AlphaFold预测所得到的数据,综合去考虑蛋白的结构特征和折叠组装等。[1] 为建立一个确凿可靠的基准(baseline),我们开发了Fidabio PDB (Protein Data Bank) Correlator软件。在下面的例子中,我们用这个软件去预测牛β-乳球蛋白(β-lactoglobulin)、人血清白蛋白、牛血清白蛋白和鸡溶菌酶(lysozyme)在溶解状态下的Rh(图2);随后,用配有紫外检测器的Fida1设备测定无标记时这些蛋白的Rh值,并与前述预测值进行比较。实验过程如图1所示。
实验表明,我们在结构数据和溶液中Rh测量值之间建立了精确的关联,本方法亦可对AlphaFold[1]预测的结构结果进行验证。此外,Fidabio PDB Correlator也适用于蛋白有荧光标记时的检测。
图1:Fidabio PDB Correlator工作流程。
FIDA测量原理
FIDA原理:流动诱导分散分析(Flow Induced Dispersion Analysis,FIDA)是一种基于毛细管的微流路检测方法。毛细管中心的流速比边缘快,因此流体在扩散的前端和尾部产生出径向浓度梯度,通过检测其中指示剂(indicator)的荧光信号即可实现“第一原理”的分子生物物理大小尺寸的测量(拓澳编者注:“第一原理”指检测所得为绝对值,意味着无需标准品做对照或需要设备校准,更多原理介绍请参考往期内容),FIDA目前已广泛应用于生物分子稳定性、相互作用等方面的研究。欲了解更多信息,请参考文末“往期链接”。
材料与方法
Fida 1仪器,Fidabio 280nm LED荧光进行尺寸大小检测(Fida Biosystems ApS)。Fidabio标准毛细管(i.d.:75µm,LT:100cm,Leff:84 cm)。
牛β-乳球蛋白(BLG)、人血清白蛋白(HSA)、牛血清白蛋白(BSA)和鸡溶菌酶(LYZ)分别在缓冲液(1× PBS)中溶解作为指示剂(Indicator)。空白缓冲液则作为分析物(Analyte)。
样品分析:先在毛细管中充满分析物,然后注入经预孵育的指示剂,在400mbar压力下向检测器移动。其中,检测LYZ的毛细管先用HS试剂(Fida Biosystems ApS)做包被。
利用Fidabio PDB Correlator将测得的流体力学半径(Rh)与分子量进行关联。相关晶体结构来自蛋白质数据库(Protein Data Bank,https://www.rcsb.org/)。用PyMOL2.5.0分子三维结构显示软件 (Schrödinger LCC)去除结晶水和共结晶分子,再用该软件做PDB文件清理和可视化(clean up and visualization)。数据分析用Fida软件进行。所有的标准偏差来自9次重复实验的误差。
结果
测量无标记时的Rh,及用利用Fidabio PDB Correlator做结构关联
配有紫外检测器的Fidabio 1仪器能检测无标记时的流体力学半径(Rh)绝对值。分别测定牛β-乳球蛋白(BLG)、人血清白蛋白(HSA)、牛血清蛋白(BSA)和鸡溶菌酶(LYZ)的尺寸大小。已知BLG在pH中性时为同源二聚体,其他pH条件下则为单体蛋白[2-5]。利用Fidabio PDB Correlator,将PDB数据加载到Fida软件中,然后预测其分子量和Rh。对于大的单体蛋白,预测所得分子量与理论值的差异在2%以内(表1和附录)。对于较小的蛋白,当其包含有共结晶的分子结构时,则建议对PDB文件先做Clean up,图2展示了用PyMOL做结构Clean up时的步骤。

表1:几种蛋白的流体力学半径(Rh)的预测值(Predicted Rh)和测量值(Apparent Rh),分子量(m.w.)的预测值和理论值,以及在PDB中的编码。预测值是将Celan up后的PDB文件加载到Fidabio PDB Correlator中生成。测量值则由Fida1测得,误差为9个重复的标准偏差。亦可见附录。
不做Clean up时,LYZ、BSA和HSA的表观Rh与实验Rh的差异均在10%以内,移除共结晶分子后,精度则可达0.3%(理论值与实验数据比值)(表1)。其中BLG的Rh预测值为2.19nm,但实验值却大了28%(附录,表1),提示其在溶解状态时存在的结构并非单体。将该文件做Clean up并选用一种同型二聚体结构,这样再预测所得的Rh为2.85 nm,与实验值2.77±0.06nm相比,差异在2.8%以内。这表明所测的BLG确为同源二聚体,而这在其他文献中已经被广泛证实[2]。
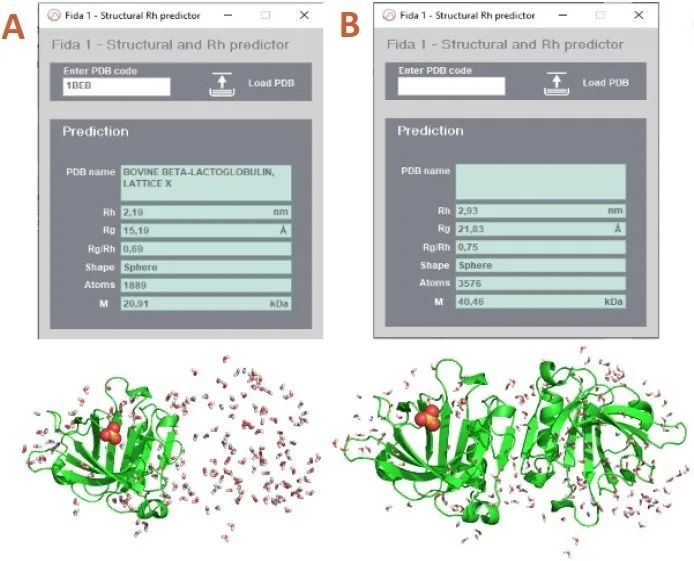
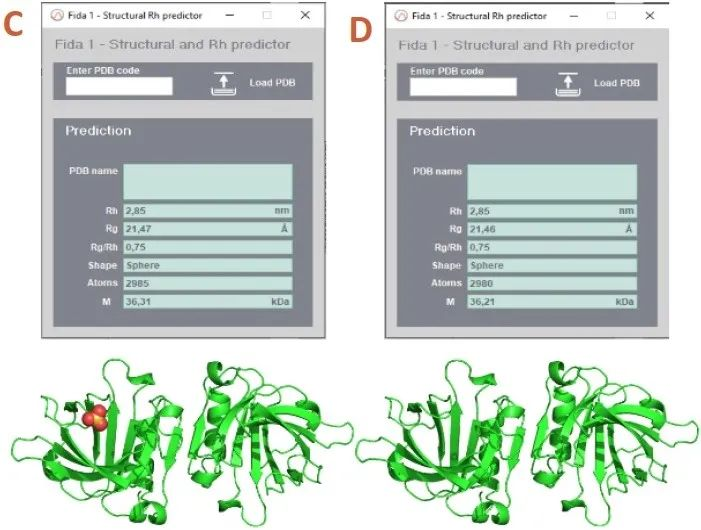
图2:Fidabio PDB Correlator和用于关联的可视化PDB文件。(A)使用Fidabio PDB Correlator直接从PBD加载BLG(1BEB) PDB文件;(B)使用PyMOL分子可视化系统生成的PDB文件;(C)去除结晶水的PDB文件;(D)去除共结晶分子并用正确的寡聚体状态做Clean up。在本例的特殊情况下,去除共结晶分子后Rh没有出现差异(在C和D两步中,Rh都是2,85nm)。然而,对于许多含有较大的共结晶分子的晶体结构,去除共结晶分子是一个重要的步骤,否则会影响Rh的预测准确性。
结论
实验结果表明,用非常少量的无标记样品(~40 nL/run at 0.1 g/L),Fida 1就能在溶液中检测出给定蛋白的寡聚状态和流体力学半径。用Fidabio PDB Correlator,则可在蛋白的流体力学半径与其结构数据或AlphaFold预测的结构数据间建立精确的关联(误差<9%)。
